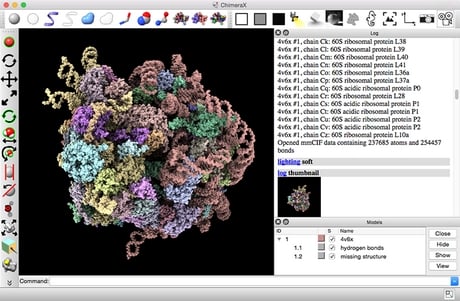
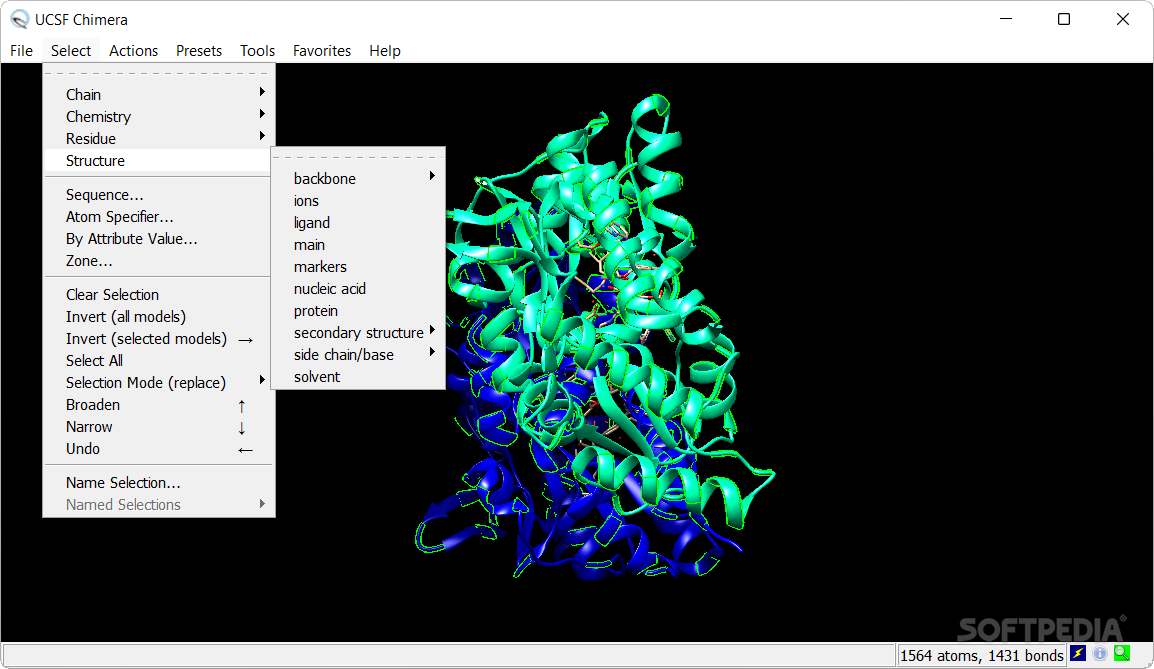
UCSF Chimera (o simplemente Chimera) es un programa extensible para la visualización interactiva y el análisis de estructuras moleculares y datos relacionados, incluyendo mapas de densidad, ensamblajes supramoleculares, alineaciones de secuencias, resultados de docking, trayectorias y conjuntos conformacionales.[1] Se pueden crear imágenes y películas de alta calidad. Chimera incluye una completa documentación y puede descargarse gratuitamente para uso no comercial.
Chimera ha sido desarrollado por el Resource for Biocomputing, Visualization, and Informatics (RBVI) de la Universidad de California, San Francisco. El desarrollo está parcialmente apoyado por los Institutos Nacionales de Salud. El programa de próxima generación es UCSF ChimeraX.[2]
Junto con swissdock, ha sido usado para simulación de acoplamiento proteína-ligando, lo que es esencial para el descubrimiento de nuevas dianas;[3] también ha sido usado para visualizar superficies de microcopía de fluorescencia,[4] y, integrado en servicios web, permite un conjunto ampliado de funciones, incluida la realización de tareas de cálculo intensivo y búsquedas en bases de datos.[5]
Véase también
- Wikimedia Commons alberga una categoría multimedia sobre UCSF Chimera.
- Lista de sistemas de gráficos moleculares
- Modelado molecular
- Gráficos moleculares
- Dinámica molecular
- Editor de moléculas
Referencias
Enlaces externos
- Página web oficial
- Galería de imágenes y Galería de animaciones de Chimera
- Publicaciones sobre Chimera
- Universidad de California, San Francisco